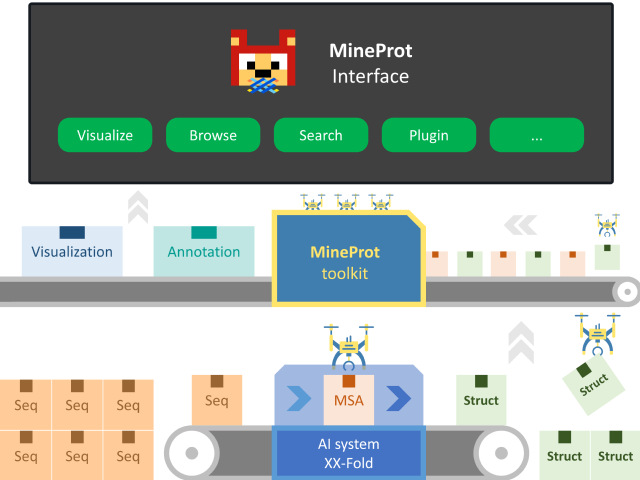
AlphaFold2等AI系统的应用极大提高了结构蛋白质组的通量。截至2022年七月,AFDB(AlphaFold Protein Structure Database)已收录超过两亿个蛋白质结构,仅用不到一年就超过了PDB数据库五十年积累的一千倍。ColabFold等改进版本的发布则进一步降低了高精度蛋白质结构预测的准入门槛。在此类AI系统的帮助下,蛋白质结构数据规模正不断扩大,其势有如当年NGS技术引发的测序数据爆炸,对传统结构数据管理应用造成了巨大压力。ModelArchive等公共存储库尚未完成针对大规模结构预测的成熟收录方案,而对多数研究者来说,开发一个类似AFDB的服务用以即时开放数据是颇有难度的。
在此背景下,东南大学生物科学与医学工程学院博士生祝云篪等开发了独立服务应用MineProt(https://github.com/huiwenke/MineProt)。该应用可帮助研究者快速搭建附带交互界面的结构蛋白质组学服务器,同时提供几乎开箱即用的脚本工具集,实现对类AlphaFold系统产生数据的自动化注释、管理与策展,具有部署便捷、功能扩展性强、用户友好等优点,有助于提高研究者生产力、促进数据分享。该成果正式出版于Database: The Journal of Biological Databases and Curation(https://doi.org/10.1093/database/baad059),并于2023年8月23日被RDMkit(The ELIXIR Research Data Management Kit)收录,成为当下结构生物信息学数据策展的推荐方案之一(https://rdmkit.elixir-europe.org/structural_bioinformatics)。
MineProt图形摘要
作为ELIXIR-CONVERGE项目的一部分,RDMkit旨在指导生命科学家按照FAIR原则更好地管理研究数据,被“地平线欧洲”计划推荐为“生命科学领域数据管理指南与良好实践的资源”。RDMkit也为诸多国际学术期刊的数据管理政策提供了参考。
东南大学生物科学与医学工程学院博士生祝云篪、硕士生童澄达和赵作翰为软件开发合作者、论文并列第一作者,陆祖宏教授为论文通讯作者。
工具信息:https://bio.tools/mineprot

